1) Academician V.I. Kulakov National Medical Research Center of Obstetrics, Gynecology, and Perinatology,
Ministry of Health of Russia, Moscow;
2) N.K. Koltsov Institute of Developmental Biology, Russian Academy of Sciences, Moscow
Objective. To study a DNA methylation profile in the placenta in fetuses with fetal growth restriction.
Subjects and methods. Thirty-eight placental samples, obtained from patients after spontaneous and surgical delivery, were investigated during the study. The women were divided into 2 groups: 1) 18 patients with a confirmed diagnosis of fetal growth restriction; 2) 20 patients with physiological pregnancy. DNA was isolated from tissues using K-sorb columns (Synthol, Russia). Then bisulfite conversion and polymerase chain reaction with primers to an island methylation fragment of the studied genes were carried out. The methylation level was determined by methylation-specific high resolution melting curve analysis using Precision Melt Analysis Software (BioRad, USA).
Results. The relative level of methylation of the TLR2 gene in the placentas in the presence of fetal growth restriction was found to be significantly lower than that in the physiological pregnancy group (p = 0.01). The study of methylation of the IGF2/H19 imprinting control region (ICR) also showed a significant decrease in the relative level of methylation in the placentas in fetal growth restriction compared with the comparison group (p <0.001).
Conclusion. The findings indicate that methylation of the TLR2 gene and the IGF2/H19 ICR play a role in fetal growth restriction and that further investigations of the levels of methylation of these genes in other biological substrates are promising in developing new diagnostic techniques.
fetal growth restriction
epigenetics
DNA methylation
placenta
В настоящее время большое значение приобретает изучение концепции фетального программирования, определяющего сформированную еще во внутриутробном периоде восприимчивость организма к заболеваниям [1–3]. Гипотеза, впервые выдвинутая D. Barker, предполагает, что неблагоприятные факторы окружающей среды вызывают морфологические и физиологические изменения у плода, адаптационный характер которых направлен на поддержание его гомеостаза [4]. В течение последних 30 лет в мировой литературе появилось множество работ о взаимосвязи между дефицитом массы тела при рождении и риском развития метаболических, сердечно-сосудистых и неврологических заболеваний [5–7]. Несмотря на понимание важности механизмов, лежащих в основе внутриутробного программирования, они остаются малоизученными [8, 9]. Согласно последним исследованиям, предположительную роль играют эпигенетические изменения некоторых генов, возникающие в ответ на негативные факторы пренатального периода [10–13]. Плацента – уникальный орган, позволяющий изучить взаимодействие генетических факторов и факторов окружающей среды, а также их вклад в развитие заболеваний [14, 15]. Изучение эпигенетических изменений ДНК плаценты представляет большой интерес в связи с перспективностью разработки новых диагностических тестов на основании определения эпигенетических меток в других биологических субстратах. Полученные знания об эпигенетической регуляции позволят в будущем внедрить их в развитие новых терапевтических методов. Одним из наиболее изученных эпигенетических механизмов, осуществляющих контроль генной экспрессии, является метилирование в области CpG динуклеотидов в промоторных регионах ДНК [16, 17].
Целью исследования было изучение профиля метилирования ДНК в плаценте при задержке роста плода (ЗРП).
Материалы и методы
Было изучено 38 образцов плацент от пациенток, которые поступили и были родоразрешены в ФБГУ «НМИЦ АГП им. В.И. Кулакова» Минздрава России. Женщины были разделены на 2 группы: 1-ю группу составили 18 пациенток c подтвержденным диагнозом ЗРП, 2-ю группу – 20 женщин с физиологическим течением данной беременности. Данное исследование было одобрено локальным этическим комитетом ФБГУ «НМИЦ АГП им. В.И. Кулакова» Минздрава России, всеми пациентками было подписано информированное согласие на участие. Критериями включения в исследование послужили: спонтанная одноплодная беременность, отсутствие острых инфекционных заболеваний, тяжелых экстрагенитальных патологий и миомы матки больших размеров у матери, отсутствие хромосомных и генетических аномалий и пороков развития у плода.
Забор плацентарной ткани производили сразу после родов. Образцы плацент были обработаны и отмыты от материнской крови в физиологическом растворе при температуре –4 °С. В исследовании были использованы ворсины хориона в месте прикрепления пуповины. Образцы тканей хранили при температуре –80° С. ДНК из ткани выделялась с использованием колонок «К-сорб» («Синтол», Россия) согласно протоколу изготовителя. Далее ДНК подвергалась бисульфитной конверсии с использованием набора Epijet (Thermo Scientific, США), в результате которой все неметилированные цитозины конвертировались в урацилы, а метилированные – не изменялись. После этого проводили полимеразную цепную реакцию (ПЦР) с праймерами к фрагменту островка метилирования генов-кандидатов, в результате которого урацилы заменялись на тимины. Отбор праймеров производили по критерию отсутствия динуклеотидов CpG. Уровень метилирования исследуемого фрагмента определяли с помощью анализа кривых плавления с высоким разрешением (methylation-specific high resolution melts, MSHRM) с использованием программного обеспечения Precision Melt Analysis Software, версия 3 (BioRad, США) [18]. Для количественной оценки метилирования использовали относительные единицы интенсивности флуоресценции (RFU) при температуре максимального расхождения кривых согласно методике, описанной ранее Красным А.М. и соавт. [19]. ПЦР и анализ кривых плавления с высоким разрешением проводили с использованием амплификатора CFX-96.
На основании данных литературы, с целью исследования уровня метилирования были отобраны гены с потенциальной ролью в патогенезе ЗРП – CDO1, MMP2, CEBPA, LEP, HLAG, VEGF, MEST, CDH1, GNA12, TLR2, BMP6, DAPK3, DFNA5, импринтинг-контролирующей области IGF2/H19.
Для статистического анализа и построения графиков использовали пакеты статистических программ Statistica 10 и OriginPro 8.5 (США). Для проверки гипотезы о нормальном распределении использовался критерий Шапиро–Уилка, для проверки равенства дисперсий – критерий Левина. Статистический анализ проводили с помощью параметрического t-критерия Стьюдента при соблюдении нормального распределения и непараметрического критерия Манна–Уитни – при несоблюдении условий нормального распределения. Сравнение групп по качественным признакам проводили с помощью точного критерия Фишера. Количественные данные представлены в виде медианы, верхней и нижней квартили Me (Q1;Q3). Различия между статистическими величинами считали статистически значимыми при уровне достоверности p<0,05.
Результаты и обсуждение
 Между исследуемыми группами не были получены статистически значимые различия по возрасту и антропометрическим данным между исследуемыми группами беременных (p>0,05). Гестационный срок при родоразрешении в группе с ЗРП составил 35,4 (33,7; 37,5) недели, что было обусловлено наличием показаний для экстренного досрочного родоразрешения путем операции кесарева сечения (КС). Клинико-анамнестические данные представлены в таблице.
Между исследуемыми группами не были получены статистически значимые различия по возрасту и антропометрическим данным между исследуемыми группами беременных (p>0,05). Гестационный срок при родоразрешении в группе с ЗРП составил 35,4 (33,7; 37,5) недели, что было обусловлено наличием показаний для экстренного досрочного родоразрешения путем операции кесарева сечения (КС). Клинико-анамнестические данные представлены в таблице.
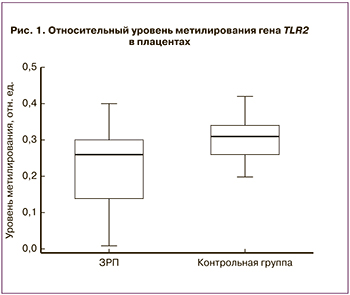
Изучение метилирования генов в плаценте показало, что относительный уровень метилирования гена толл-подобного рецептора 2 (TLR2) в группе ЗРП составил 0,26 (0,14;0,28), что было статистически ниже по сравнению с группой условно здоровых беременных (0,31 (0,26;0,34), р=0,01). Данные представлены на рис. 1.
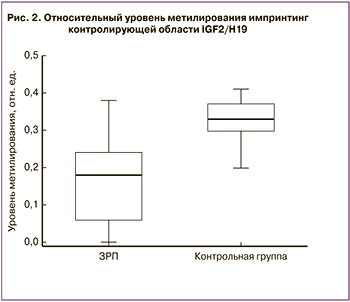
Исследование уровня метилирования импринтинг-контролирующей области инсулиноподобного фактора роста 2 (IGF2) и H19 также показало статистически значимое снижение относительного уровня метилирования в плацентах с ЗРП (0,18 (0,06;0,21)) по сравнению с контрольной группой (0,33 (0,29;0,36), p<0,001). Данные представлены на рис. 2.
 Как известно, TLR2 является мембранным белком из класса толл-подобных рецепторов, играющим ключевую роль во врожденном иммунитете, и обнаруживается в плаценте с ранних сроков беременности, увеличиваясь в присутствии инфекционных агентов [20, 21]. Кроме того, TLR2 участвует в регуляции апоптоза клеток цитотрофобласта с I триместра беременности путем усиления его активности [22–24]. Таким образом, снижение уровня метилирования гена TLR2 может приводить к усилению экспрессии TLR2 и, как следствие, повышенному апоптозу в плаценте, играющему важную роль в патогенезе ЗРП.
Как известно, TLR2 является мембранным белком из класса толл-подобных рецепторов, играющим ключевую роль во врожденном иммунитете, и обнаруживается в плаценте с ранних сроков беременности, увеличиваясь в присутствии инфекционных агентов [20, 21]. Кроме того, TLR2 участвует в регуляции апоптоза клеток цитотрофобласта с I триместра беременности путем усиления его активности [22–24]. Таким образом, снижение уровня метилирования гена TLR2 может приводить к усилению экспрессии TLR2 и, как следствие, повышенному апоптозу в плаценте, играющему важную роль в патогенезе ЗРП.
Также в плаценте наблюдается повышенная экспрессия импринтированных генов, которые важны для ее правильного развития [25–27]. В настоящее время большой интерес вызывает изучение импринтинг-контролирующей области генов IGF2 и H19 на хромосоме 11 отцовского происхождения в связи с данными о зависимости метилирования данной области с развитием ЗРП [28]. Метилирование данного региона определяет экспрессию импринтированных генов IGF2 и H19; в случае метилирования повышается экспрессия IGF2 [29]. В исследовании Koukoura O. и соавт. [30] также было продемонстрировано снижение метилирования данной области и повышение экспрессии гена H19 в плацентах при ЗРП. Большой интерес представляет изучение IGF2 в связи с его участием в регуляции процессов пролиферации, дифференцировки и транспорта питательных веществ в плаценте [31]. Xiao X. и соавт. провели исследование уровня метилирования IGF2 в плаценте путем бисульфитного пиросеквенирования и обнаружили его снижение при ЗРП [32]. В подобном исследовании St-Pierre J. и соавт. была обнаружена отрицательная корреляция между уровнем метилирования IGF2 и массой новорожденных [33]. Таким образом, полученные нами данные согласуются с мировыми и подтверждают роль IGF2 в формировании ЗРП.
Заключение
Анализ уровня метилирования исследованных генов показал статистически значимое снижение уровня метилирования гена TLR2 и импринтинг-контролирующей области IGF2/H19 в плацентах женщин с ЗРП. Полученные результаты указывают на роль метилирования гена TLR2 и импринтинг- контролирующей области IGF2/H19 в формировании задержки роста плода и перспективность дальнейшего изучения уровня метилирования данных генов в других биологических субстратах с целью внедрения новых методов диагностики.
- Barker D.J.P., Osmond C. Infant mortality, childhood nutrition, and ischaemic heart disease in England and Wales. Lancet. 1986; 10, 1(8489): 1077–81. doi: 10.1016/s0140-6736(86)91340-1
- Kwon E.J., Kim Y.J. What is fetal programming: a lifetime health is under the control of in utero health. Obstet Gynecol Sci. 2017; 60(6): 506–19. doi: 10.5468/ogs.2017.60.6.506
- Hales C.N., Barker D.J.P. Type 2 (non-insulin-dependent) diabetes mellitus: the thrifty phenotype hypothesis. Int J Epidemiol. 2013; 42(5): 1215–22 doi: 10.1093/ije/dyt133
- Дегтярева Е.И., Григорян О.Р., Волеводз Н.Н., Андреева Е.Н., Клименченко Н.И., Мельниченко Г.А., Дедов И.И., Сухих Г.Т. Роль импринтинга генов при внутриутробной задержке роста плода. Акушерство и гинекология. 2015; 12: 5–10. [Degtyareva E.I., Grigoryan O.R., Volevodz N.N., Andreeva E.N., Klimenchenko N.I., Melnichenko G.A., Dedov I.I., Sukhikh G.T. Role of gene imprinting in intrauterine growth restriction. Akusherstvo i ginekologiya/Obstetrics and Gynecology. 2015; 12: 5–10.(in Russian)].
- Marciniak A., Patro-Małysza J., Kimber-Trojnar Ż., Marciniak B., Oleszczuk J., Leszczyńska-Gorzelak B. Fetal programming of the metabolic syndrome. Taiwan J Obstet Gynecol. 2017; 56(2):133–8. doi: 10.1016/j.tjog.2017.01.001
- Menendez-Castro C., Rascher W., Hartner A. Intrauterine growth restriction - impact on cardiovascular diseases later in life. Mol Cell Pediatr. 2018; 5(1): 4. doi: 10.1186/s40348-018-0082-5
- Faa G., Manchia M., Pintus R., Gerosa C., Marcialis M.A.,Fanos V. Fetal programming of neuropsychiatric disorders.Birth Defects Res C Embryo Today. 2016; 108(3): 207–223. doi: 10.1002/bdrc.21139
- Feil R., Fraga M.F. Epigenetics and the environment: emerging patterns and implications. Nat Rev Genet. 2012;13(2): 97–109. doi: 10.1038/nrg3142.
- Manolio T. A., Collins F.S., Cox N.J., Goldstein D.B., Hindorff L.A., Hunter D.J. et al. Finding the missing heritability of complex diseases.Nature. 2009; 461(7265): 747–53. doi: 10.1038/nature08494
- Salam R.A., Das J.K., Bhutta Z.A. Impact of intrauterine growth restriction on long-term health. Curr Opin Clin Nutr Metab Care. 2014;17(3): 249–54. doi: 10.1097/MCO.0000000000000051
- Chen P.Y., Ganguly A., Rubbi L., Orozco L.D., Morselli M., Ashraf D., et al. Intrauterine calorie restriction affects placental DNA methylation and gene expression. Physiol Genomics. 2013; 45(14): 565–76. doi: 10.1152/physiolgenomics.00034.2013
- Banister C.E., Koestler D.C., Maccani M.A., Padbury J.F., Houseman E.A., Marsit C.J. Infant growth restriction is associated with distinct patterns of DNA methylation in human placentas. Epigenetics. 2011; 6(7): 920–7. doi: 10.4161/epi.6.7.16079
- Vaiman D. Genes, epigenetics and miRNA regulation in the placenta. Placenta. 2017; 52:127–33. doi: 10.1016/j.placenta.2016.12.026
- Marsit C.J. Placental epigenetics in children’s environmental health. Semin Reprod Med. 2016; 34(1): 36–41. doi:10.1055/s-0035-1570028
- Lillycrop K.A., Burdge G.C. Environmental challenge, epigenetic plasticity and the induction of altered phenotypes in mammals. Epigenomics. 2014; 6(6): 623–36. doi: 10.2217/epi.14.51
- Nelissen E.C.M., van Montfoort A.P., Dumoulin J.C., Evers J.L. Epigenetics and the placenta. Hum Reprod Update. 2011;17(3): 397–417. doi: 10.1093/humupd/dmq052
- Moore L.D., Le T., Fan G. DNA methylation and its basic function. Neuropsychopharmacology. 2013; 38(1): 23–38. doi: 10.1038/npp.2012.112
- Wojdacz T.K., Dobrovic A., Hansen L.L. Methylation-sensitive high-resolution melting. Nat Protoc. 2008 3(12):1903–8. doi: 10.1093/nar/gkm013
- Красный А.М., Садекова А.А., Волгина Н.Е., Машаева Р.И., Кометова В.В., Хабас Г.Н., Голицына Ю.С., Носова Ю.В., Оводенко Д.Л. Исследование уровня метилирования гена RASSF1 в плазме и опухоли при раке эндометрия. Бюллетень экспериментальной биологии и медицины. 2019; 2: 223–7. [Krasnyi A.M., Sadekova A.A., Volgina N.E., Mashaeva R.I., Kometova V.V., Khabas G.N., Golitsyna Yu.S., Nosova Yu.V., Ovodenko V.L.. Investigation of the level of methylation of the RASSF1 gene in plasma and tumor in endometrial cancer. Bulletin of Experimental Biology and Medicine. 2019; (2): 223–7. (in Russian)]
- Ma Y., Krikun G., Abrahams V.M., Mor G., Guller S. Cell type-specific expression and function of toll-like receptors 2 and 4 in human placenta: implications in fetal infection. Placenta. 2007; 28(10): 1024–31. doi: 10.1016/j.placenta.2007.05.003
- Erboga M., Kanter M. Trophoblast cell proliferation and apoptosis in placental development during early gestation period in rats. Anal Quant Cytopathol Histpathol. 2015; 37(5): 286–94.
- Koga K., Aldo P.B., Mor G. Toll‐like receptors and pregnancy: trophoblast as modulators of the immune response. J Obstet Gynaecol Res. 2009; 35(2): 191–202. doi: 10.1111/j.1447-0756.2008.00963.x
- Abrahams V.M., Bole-Aldo P., Kim Y.M., Straszewski-Chavez S.L., Chaiworapongsa T., Romero R., et al. Divergent trophoblast responses to bacterial products mediated by TLRs. J Immunol. 2004; 173(7): 4286–96. doi: 10.4049/jimmunol.173.7.4286
- Silva J.F., Ocarino N.M., Serakides R. Spatiotemporal expression profile of proteases and immunological, angiogenic, hormonal and apoptotic mediators in rat placenta before and during intrauterine trophoblast migration. Reprod Fertil Dev. 2017; 29(9): 1774–86. doi:10.1071/RD16280
- Tycko B. Imprinted genes in placental growth and obstetric disorders. Cytogenet Genome Res. 2006; 113(1–4): 271–8. doi: 10.1159/000090842
- John R.M. Imprinted genes and the regulation of placental endocrine function: Pregnancy and beyond. Placenta. 2017; 56: 86–90. doi: 10.1016/j.placenta.2017.01.099
- Christians J.K., Leavey K., Cox B.J. Associations between imprinted gene expression in the placenta, human fetal growth and preeclampsia. Biol Lett. 2017;13(11): pii: 20170643.doi: 10.1098/rsbl.2017.0643
- Bartholdi D., Krajewska-Walasek M., Ounap K., Gaspar H., Chrzanowska K.H., Ilyana H. et al. Epigenetic mutations of the imprinted IGF2-H19 domain in Silver–Russell syndrome (SRS): results from a large cohort of patients with SRS and SRS-like phenotypes. J Med Genet. 2009; 46(3): 192–7. doi: 10.1136/jmg.2008.061820
- Du M., Zhou W., Beatty L.G., Weksberg R., Sadowski P.D., et al. The KCNQ1OT1 promoter, a key regulator of genomic imprinting in human chromosome 11p15. 5. Genomics. 2004; 84(2): 288–300. DOI: 10.1016/j.ygeno.2004.03.008
- Koukoura O., Sifakis S., Soufla G., Zaravinos A., Apostolidou S., Jones A., et al. Loss of imprinting and aberrant methylation of IGF2 in placentas from pregnancies complicated with fetal growth restriction. Int J Mol Med. 2011; 28(4): 481–7. doi: 10.3892/ijmm.2011.754.
- Tabano S., Colapietro P., Cetin I., Grati F.R., Zanutto S., Mandò C., Antonazzo P. et al. Epigenetic modulation of the IGF2/H19 imprinted domain in human embryonic and extra-embryonic compartments and its possible role in fetal growth restriction. Epigenetics. 2010; 5(4): 313–24. DOI: 10.4161/epi.5.4.11637
- Xiao X., Zhao Y., Jin R., Chen J., Wang X., et al. Fetal growth restriction and methylation of growth-related genes in the placenta. Epigenomics. 2016; 8(1): 33–42. doi: 10.2217/epi.15.101.
- St-Pierre J., Hivert M.F., Perron P., Poirier P., Guay S.P., Brisson D., et al. IGF2 DNA methylation is a modulator of newborn’s fetal growth and development. Epigenetics. 2012; 7(10): 1125–32. doi: 10.4161/epi.21855.
Received 20.06.2019
Accepted 21.06.2016
Zarine V. Khachatryan, postgraduate student of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.). Tel.: +7-909-656-24-56. E-mail:
z.v.khachatryan@gmail.com
Natalia E. Kan, PhD, MD, professor of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.). Tel.: +7-926-220-86-55. E-mail:
kan-med@mail.ru.
Number Researcher ID B-2370-2015. ORCID ID 0000-0001-5087-5946
Aleksey M. Krasnyi, PhD, the head of the cytology laboratory of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named
after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.), senior scientist, laboratory of evolutionary developmental biology,
N.K. Koltzov Institute of Developmental Biology of Russian Academy of Sciences (119334, Moscow, Vavilova str. 26, Russia. Tel.: +7-495-438-22-72. E-mail:
alexred@list.ru
Alsu A. Sadekova, PhD, scientific researcher of the cytology laboratory of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.). Tel.: +7-495-438-22-72. E-mail:
a_sadekova@oparina4.ru
Sergey V. Kurevlev, scientific researcher of the cytology laboratory of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.). Tel.: +7-985-693-66-33. E-mail:
s_kurevlev@oparina4.ru
Victor L. Tyutyunnik, PhD, MD, professor of the National Medical Research Center for Obstetrics, Gynecology and Perinatology named after Academician V.I. Kulakov Ministry of Health of Russia (117997, Moscow, Ac. Oparina, 4 str.). Tel.: +7-903-969-50-41. E-mail:
tioutiounnik@mail.ru.
Number Researcher ID B-2364-2015.ORCID ID 0000-0002-5830-5099
For citation: Khachatryan Z.V., Kan N.E., Krasnyi A.M., Sadekova A.A., Kurevlev S.V., Tyutyunnik V.L. Gene methylation in the placenta of fetuses with fetal growth restriction.
Akusherstvo i Ginekologiya /Obstetrics and Gynecology. 2019; (12): 54-8. (in Russian).
http://dx.doi.org/10.18565/aig.2019.12.54-58
 Между исследуемыми группами не были получены статистически значимые различия по возрасту и антропометрическим данным между исследуемыми группами беременных (p>0,05). Гестационный срок при родоразрешении в группе с ЗРП составил 35,4 (33,7; 37,5) недели, что было обусловлено наличием показаний для экстренного досрочного родоразрешения путем операции кесарева сечения (КС). Клинико-анамнестические данные представлены в таблице.
Между исследуемыми группами не были получены статистически значимые различия по возрасту и антропометрическим данным между исследуемыми группами беременных (p>0,05). Гестационный срок при родоразрешении в группе с ЗРП составил 35,4 (33,7; 37,5) недели, что было обусловлено наличием показаний для экстренного досрочного родоразрешения путем операции кесарева сечения (КС). Клинико-анамнестические данные представлены в таблице. Как известно, TLR2 является мембранным белком из класса толл-подобных рецепторов, играющим ключевую роль во врожденном иммунитете, и обнаруживается в плаценте с ранних сроков беременности, увеличиваясь в присутствии инфекционных агентов [20, 21]. Кроме того, TLR2 участвует в регуляции апоптоза клеток цитотрофобласта с I триместра беременности путем усиления его активности [22–24]. Таким образом, снижение уровня метилирования гена TLR2 может приводить к усилению экспрессии TLR2 и, как следствие, повышенному апоптозу в плаценте, играющему важную роль в патогенезе ЗРП.
Как известно, TLR2 является мембранным белком из класса толл-подобных рецепторов, играющим ключевую роль во врожденном иммунитете, и обнаруживается в плаценте с ранних сроков беременности, увеличиваясь в присутствии инфекционных агентов [20, 21]. Кроме того, TLR2 участвует в регуляции апоптоза клеток цитотрофобласта с I триместра беременности путем усиления его активности [22–24]. Таким образом, снижение уровня метилирования гена TLR2 может приводить к усилению экспрессии TLR2 и, как следствие, повышенному апоптозу в плаценте, играющему важную роль в патогенезе ЗРП.



